
Выделение и идентификация дрожжей перспективных для утилизации лактозы
с лактозоутилизирующими свойствами. В основном дрожжевые культуры были выделены из молочнокислых продуктов домашнего приготовления различных регионов страны. Дрожжи обладают характерными морфо-культуральными признаками. Идентификация 19 дрожжей проведена биохимическим методом и методом генотипирования с помощью ПЦР анализа ITS региона. Роды дрожжей представлены Kluyveromyces, Candida, Saccharomyces, являющимися перспективными в качестве утилизации лактозы.
Лактозосбраживающие дрожжи, играющие важную роль в молочной промышленности (к ним относятся роды Torulopsis, Kluyveromyces и др.) при приготовлении кефира и кумыса, сбраживают молочный сахар, образуя спирт и углекислоту, вследствие чего улучшается вкус продукта и повышается его усвояемость организмом [3]. Важнейшими для молочного производства являются лактозосбраживающие Saccharomyces lactis. Они используются для приготовления кефира (из коровьего молока) и кумыса (из кобыльего молока). Известен способ производства кумысного продукта на основе молочной сыворотки с использованием живых клеток дрожжей [1].
Дрожжи, сбраживающие лактозу, были идентифицированы Скородумовой А.М. как Fabospora fragilis, Saccharomyces lactis, Zygosaccharomyces lactis. Способностью сбраживать лактозу обладают также некоторые виды Candida и Torulopsis – Candida pseudotropicalis var. lactosa, Torulopsis kefir, Torylopsis sphaerica, выделенные из кефирного грибка [5].
Применение культур дрожжей с известными свойствами дает возможность эффективнее использовать их деятельность – экономичнее перерабатывать сырье, получать высокий выход и надлежащее качество продукции [2].
Для идентификации дрожжевых культур используются классические микробиологические методы. Однако в последние годы наблюдается полифазный подход, который заключается в объединении всех возможных данных как фенотипического, так и генетического характера, с целью получения достоверной информации [1].
Целью нашей работы являлось выделение и идентификация дрожжей из различных субстратов для создания заквасок с лактозоутилизирующими свойствами.
Материалы и методы исследования
Из субстратов отбирали по 1 мл (г) и засевали в бульонную среду Сабуро производства HiMedia Laboratories Pvt (Индия). Инкубировали при 30–37 ºС – 24–48 часов. Для получения чистых культур производили десятикратные разведения и с последних разведений делали посев на чашки Петри с агаризованной средой Сабуро в количестве 0,1 мл. После инкубации для первоначальной идентификации изучали морфолого-культуральные свойства и микроскопию выросших колоний. Жизнеспособность культур микроорганизмов определяли методом Miles&Misra [4].
Видовую идентификацию дрожжей определяли биохимическим методом на автоматизированном микробиологическом анализаторе BioMerieuxVitek 2 Compact systems с применением карт YST, а также методом определения прямой нуклеотидной последовательности фрагмента ITS региона, с последующим определением нуклеотидной идентичности с последовательностями, депонированными в международной базе данных GeneBank.
Концентрацию ДНК измеряли спектрофотометрическим методом с использованием спектрофотометра NanoDrop при длине волны 260 нм [10].
Амплификация фрагмента ITS региона.
Реакция ПЦР была выполнена с [6] использованием прямого праймера ITS 1 (5’ TCC GTA GGT GAA CCT GCG G ‘3) и обратного ITS 4 (5’ TCC TCC GCT TAT TGA TAT GC ‘3), в общем объеме 20 мкл. ПЦР смесь содержала 150 нг ДНК, 1 Ед. MaximaHotStartTaqDNAPolymerase (Fermentas), 0,2 mM каждого дНТФ, 1-х ПЦР буфер (Fermentas), 2,5 mMMgCl2, 10 пмоль каждого праймера. Программа ПЦР амплификации включала длительную денатурацию 95 °С в течение 7 минут; 30 циклов: 95 °С – 30 секунд, 52 °С – 40, 72 °С – 1 минута; заключительная элонгация 7 минут при 72 °С, ПЦР программа была выполнена с применением амплификатора GeneAmpPCRSystem 9700 (Applied Biosystems).
Определение нуклеотидной последовательности.
Очистку ПЦР продуктов от несвязавшихся праймеров проводили ферментативным методом, используя ExonucleaseI (Fermentas) и щелочную фосфатазу (Shrimp Alkaline Phosphatase, Fermentas) [7]. Реакцию секвенирования проводили с применением BigDye® Terminator v3.1 Cycle Sequencing Kit (Applide Biosystems) согласно инструкции производителя, с последующим разделением фрагментов на автоматическом генетическом анализаторе 3730 x lDNA Analyzer (Applide Biosystems).
Анализ нуклеотидных последовательностей.
Нуклеотидные последовательности ITS региона идентифицируемых объектов были анализированы и объединены в общую последовательность в программном обеспечении SeqScape 2.6.0 (Applide Biosystems). После чего были удалены концевые фрагменты (нуклеотидные последовательности праймеров, фрагменты, имеющие низкий показатель качества) что позволило нам получить нуклеотидную последовательность протяженностью 700 п.н., которые были идентифицированы в GeneBank по алгортму BLAST.
В анализ были включены нуклеотидные последовательности ITS региона наиболее филогенетически связанных дрожжей.
Построение филогенетических деревьев с использованием программного обеспечением Mega 3.1, выравнивание нуклеотидных последовательностей проводили, используя алгоритм ClustalW, построение филогенетических деревьев проводили с использованием метода ближайших соседей (Neiighbor-Joining NJ).
Результаты исследования и их обсуждение
Проведено выделение изолятов дрожжей из кисломолочных продуктов домашнего приготовления (различных регионов страны), разных фруктов и овощей. Многие исследователи считают, что бактерии, выделенные из местных продуктов, обладают наиболее выраженными лечебными свойствами для жителей конкретной географической зоны в отличие от импортных продуктов. Было выделено 38 чистых изолятов дрожжей. Микроскопия выделенных культур представлена на рис. 1.
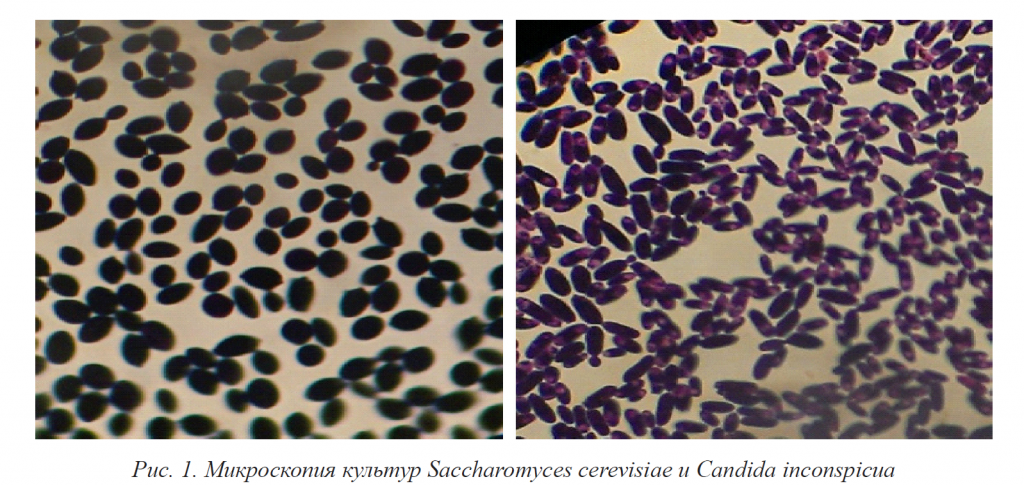
Они соответствуют морфологической характеристике: грамположительные дрожжевые клетки, круглые, овальные, лимоновидные, крупные, размеры варьируют от мелких до крупных. На твердых питательных средах Сабуро большинство культур представляли собой круглые, крупные колонии, рельеф колоний с приподнятой в виде соска серединой и валиком по периферии, поверхность блестящая с глянцем, бежевого или белого цвета, пастообразной консистенции (рис. 2).
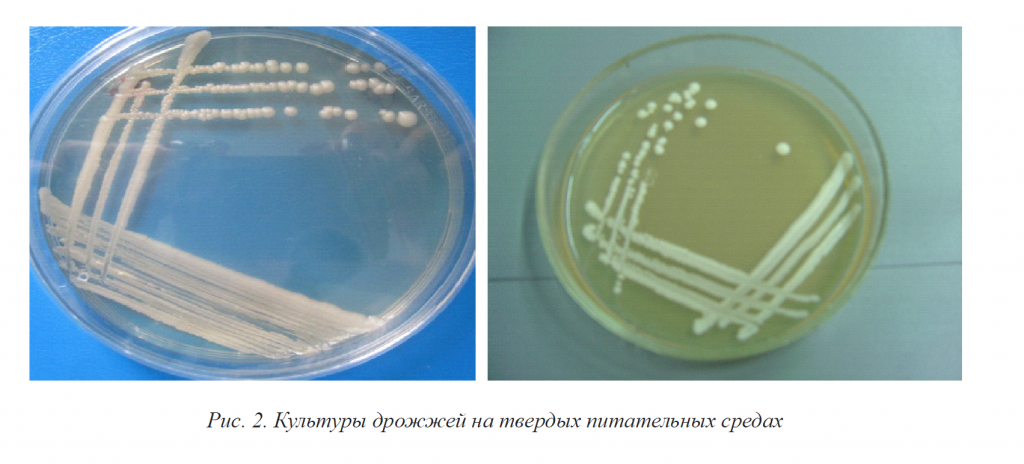
В бульонной среде рост отмечался в виде обильного осадка на дне пробирки,а также в виде пристеночного роста. Консистенция пастообразная, среда над осадком прозрачная, цвет белый, при встряхивании размешивается в виде хлопьев до равномерной мутности. Отобранные нами по показателю жизнеспособности (108–109 КОЕ/мл) культуры дрожжей в количестве 19 были идентифицированы на бактериологическом анализаторе Vitek 2 compact («BioMerieux», США, Франция) с использованием карт YST для идентификации дрожжевых и дрожжеподобных грибов. Карты состоят из 46 биохимических тестов, т.е. идентификация основана на сбраживании углеводов и многоатомных спиртов.
Из 19 исследуемых культур к Candida kefir отнесены 5 культур дрожжей, к Candida lusitaniae – 3, к Candida famata – 4, к Candida krusei – 2, к Candida lambica – 1,
к Cryptococcus laurentii – 1, а 3 культуры не идентифицированы на бактериологическом анализаторе.
При идентификации культур микроорганизмов только с использованием стандартных микробиологических методов могут возникнуть трудности. Следовательно, необходимо использовать несколько подходов, тем более 3 культуры дрожжей не были идентифицированы на баканализаторе, несмотря на то, что по культурально-морфологическим характеристикам соответствуют дрожжевым и дрожжеподобным грибам.
В 1980-х годах в практику вошел новый стандарт идентификации бактерий, позволяющий идентифицировать микроорганизм, не имея представлений о его биологических особенностях. С. Woese с сотрудниками установили, что филогенетические отношения бактерий могут быть определены путем сравнения высококонсервативной части генетического кода, что послужило началом развития интенсивно развивающейся универсальной системы идентификации [9]. Высокая идентичность нуклеотидной последовательности ITS региона, по сравнению с другими рРНК генами, позволила использовать его как стандартный генетический маркер для идентификации и таксономической классификации бактериальных разновидностей [8]. Метод основан на определении нуклеотидной последовательности ITS региона, с достаточным количеством внутривидовых полиморфизмов, позволяющих проводить статистически достоверную идентификацию.
В нашем исследовании генетическая идентификация 19 штаммов была осуществлена методом определения нуклеотидной последовательности фрагмента ITS региона, с последующим определением нуклеотидной идентичности с последовательностями, депонированными в международной базе данных Gene Bank, а также построением филогенетических деревьев с нуклеотидными последовательностями штаммов. Процедура выделения хромосомной ДНК по методу Kate Wilson позволяет эффективно выделять ДНК грамположительных и грамотрицательных бактерий, а также ДНК грибов и дрожжей с предварительной гомогенизацией биомассы.
В результате выделения ДНК были получены образцы ДНК с высокой концентрацией хорошего качества: значение 260/280 варьирует от 1,99–2,19. Методом ПЦР был амплифицирован фрагмент ITS региона, молекулярной массой около 700 п.н. у всех идентифицируемых штаммов. Продукты ПЦР амплификации были использованы для определения нуклеотидной последовательности. Далее нуклеотидные последовательности ITS региона выделенных культур дрожжей были сравнены с наиболее филогенетически связанными штаммами дрожжей базы данных и построено филогенетическое дерево (рис. 3).
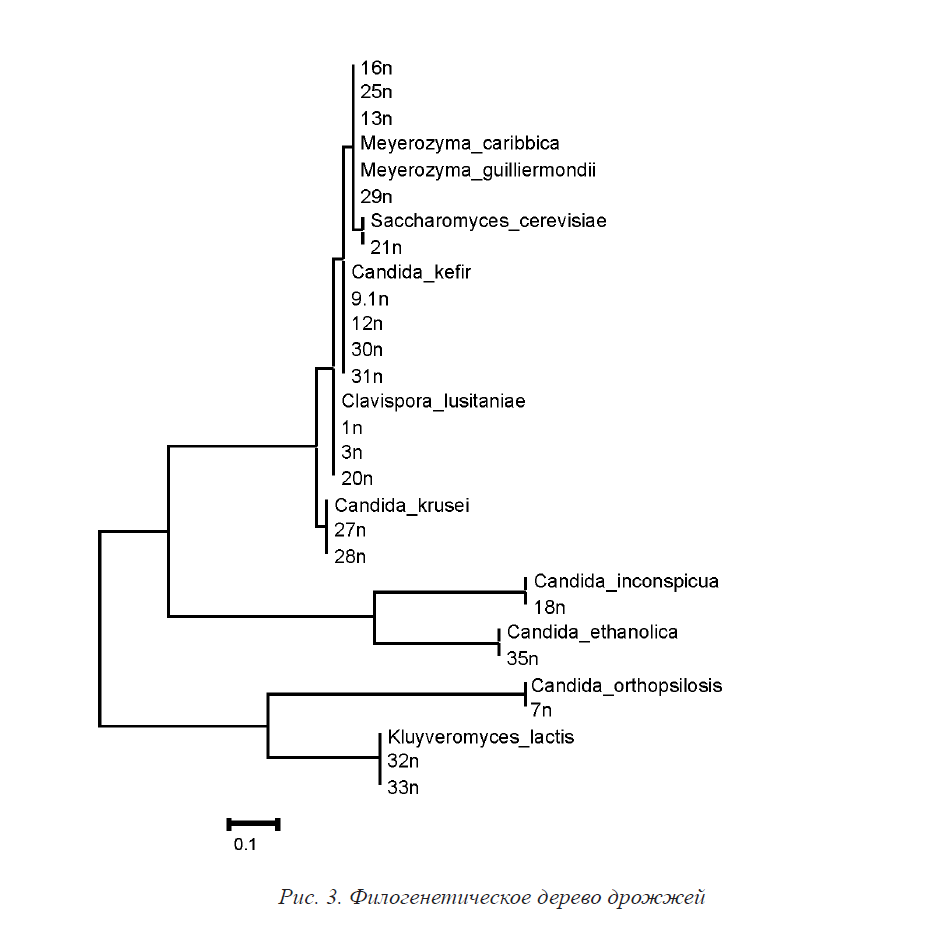
Как видно на рис. 3, штаммы 25n, 29n, 16n и 13n при филогенетическом анализе фрагмента ITS региона были объединены в один кластер с Meyerozyma guilliermondii и Meyerozyma caribbica. Штамм 21n c Saccharomyces cerevisiae, 9.1n, 12n, 30n и 31n c Candida kefir, 1n, 3n и 20n с Clavispora lusitaniae, 27n и 28n с Candida krusei, 18n c Candida inconspicua, 35n c Candida ethanolica, 7n c Candida orthopsilosis. Kluyveromyces lactis соответствует 32n и 33n. Итак, все штаммы относятся к дрожжевым культурам.
Результаты идентификации дрожжей на бактериологическом анализаторе Bio MerieuxVitek 2 Compact и методом генотипирования по фрагменту ITS региона представлены в таблице.
Результаты идентификации
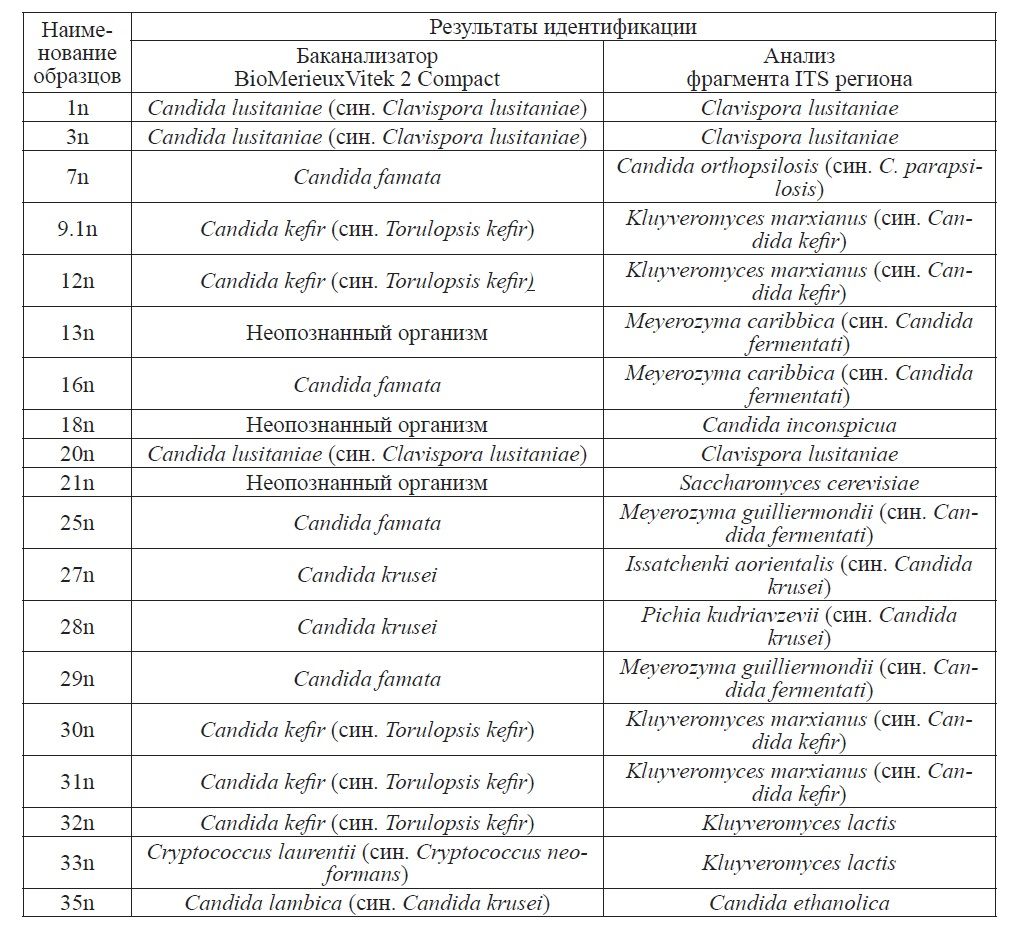
Данные, приведенные в таблице, свидетельствуют о том, что все идентифицированные культуры по двум методам подтверждают их близкородственность. Kluyveromyces marxianus является одним из видов дрожжей рода Candida kefyr. К. marxianus используется для коммерческого производства фермента лактазы. Kluyveromyces lactis (ранее назывался Saccharomyces lactis) представляет собой дрожжи, которые обладают способностью усваивать лактозу и преобразовать его в молочную кислоту.
Candida lusitaniae, Clavispora lusitaniae, Pichia kudriavzevii, Meyerozyma caribbica Meyerozyma guilliermondii является одним из видов дрожжей в роду Candida. Candida kefyr (синоним Torulopsis kefir) и Candida krusei (синоним Issatchenkia orientalis) содержатся в обычном кефире. Таким образом, для достоверной идентификации бактерий в настоящее время недостаточно опираться на данные, полученные с использованием одного метода, так как методы имеют свои дискриминационные ограничения. Поэтому в настоящее время активно используется сочетание методов, что позволяет преимуществами одного метода компенсировать недостатки другого метода.
Заключение
В ходе проделанной работы выделено и идентифицировано 19 штаммов дрожжей с максимальным показателем жизнеспособности 108–109 КОЕ/мл. Метод секвенирования ITS региона ДНК и бактериологическом анализаторе идентификация дрожжей дает высокую результативность в определении видовой и расовой принадлежности. Исследование нуклеотидных последовательностей ITS региона полученных нами дрожжей показало 99–100 % сходство с базовым вариантом, а на баканализатаре показало 89–92 %.
На основе полученных результатов выявлено, что все выделенные культуры относятся к дрожжевым культурам, а как известно, именно данные виды обладают свойством утилизировать лактозу.
Список литературы
1. Артюхова С.И. Молочная сыворотка для производства кумысного продукта // Пищевая промышленность. – 2011. – № 5. – С. 26–27.
2. Рябцева С.А., Анисимов Г.С., Скрипнюк А.А. Дрожжи в молочной промышленности: причина порчи, нормирование, определение // Молочная промышленность. – 2013. – № 5. – С. 67–68.
3. Скородумова А.М. Дрожжи молока и молочных продуктов и их производственное значение. – М.: Пищевая промышленность, 1969. – 120 с.
4. Скородумов Д.И., Субботин В.В., Сидоров Н.А. и др. Микробиологическая диагностика бактериальных болезней животных. – М.: Изографъ, 2005. – С. 19–23.
5. Хамнаева Н.И. Кефирные грибки: использование биотехнологических свойств при производстве бактериальных заквасок. – М.: МГУПБ, 2000. – 89 с.
6. Clayton R.A., Sutton G., Hinkle P. S. et al. Intraspecific variation in small-subunit rRNA sequences in GenBank: why single sequences may not adequately represent prokaryotic taxa // International J. l of Systematic Bacteriology. – 1995. – Vol. 45. – P. 595–599.
7. Kumar S., Tamura K., Nei M. MEGA3: Integrated software for Molecular Evolutionary Genetics Analysis and sequence alignment // Briefings in bioinformatics. – 2004. –
Vol. 5, № 2. – P. 150–163.
8. Palys T., Nakamura L.K., Cohan FM. Discovery and classifi cation of ecological diversity in the bacterial world: the role of DNA sequence data // International J. of Systematic Bacteriology. – 1994. – Vol. 47. – P. 1145–1156.
9. Vandamme P., Pot B., Gillis M. et al. Polyphasic Taxonomy, a Consensus Approach to Bacterial Systematics // Microbiological Reviews. – 1996. – Vol. 60, № 2. – Р. 407–438.
10. Werle E., Schneider C., Renner M. et al. Convenient single-step, one tube purification of PCR products for direct sequencing // Nucleic Acids Res. – 1994. – Vol. 22. –
P. 4354–4355.
Источник: журнал "Успехи современного естествознания". 2015. № 2. С. 126-131.

Галерея продукции
 Добавка комплексная пищевая «МОНАМИЛК» - СМЕСЬ №2 Р5Н ДЛЯ ТВОРОГА И ТВОРОЖНОГО ПРОДУКТА
Добавка комплексная пищевая «МОНАМИЛК» - СМЕСЬ №2 Р5Н ДЛЯ ТВОРОГА И ТВОРОЖНОГО ПРОДУКТА
 Саморазгружающийся сепаратор-концентратор для получения высокожирных сливок - ВЖС Frautech Separators (Италия)
Саморазгружающийся сепаратор-концентратор для получения высокожирных сливок - ВЖС Frautech Separators (Италия)
 Смесь токоферолов 70% (жидкость)
Смесь токоферолов 70% (жидкость)
 Горизонтальная упаковочная машина ЛИНЕПАК Ф3М HS (High speed) для упаковки в пакеты Флоу-пак
Горизонтальная упаковочная машина ЛИНЕПАК Ф3М HS (High speed) для упаковки в пакеты Флоу-пак
 Система бескрановой посолки сыра
Система бескрановой посолки сыра








